中国科学家运用人工智能算法发现大量全新 RNA 病毒,大幅拓宽 RNA 病毒库
GoodNav 10 月 10 日报道,来自中山大学官方微信公众号的信息显示,10 月 9 日,中山大学医学院施莽教授团队与阿里云李兆融团队在《细胞》(Cell)杂志上发表了一项研究,揭示了 180 个超群和超过 16 万种全球 RNA 病毒的发现,这一成果是迄今为止规模最大的 RNA 病毒研究,显著丰富了全球 RNA 病毒的多样性。此次研究将人工智能技术应用于病毒鉴定,发现了传统方法忽视的病毒“暗物质”,为病毒学研究开辟了新方向。
传统病毒发现方法主要依赖于病毒分离及生命组学的生物信息学分析,这些方法高度依赖已有的知识,对 RNA 病毒的识别效率却相对较低,因其种类繁多且易变异。该研究团队开发的 LucaProt 人工智能算法能够对病毒和非病毒基因组序列进行深度学习,并独立判断病毒序列。
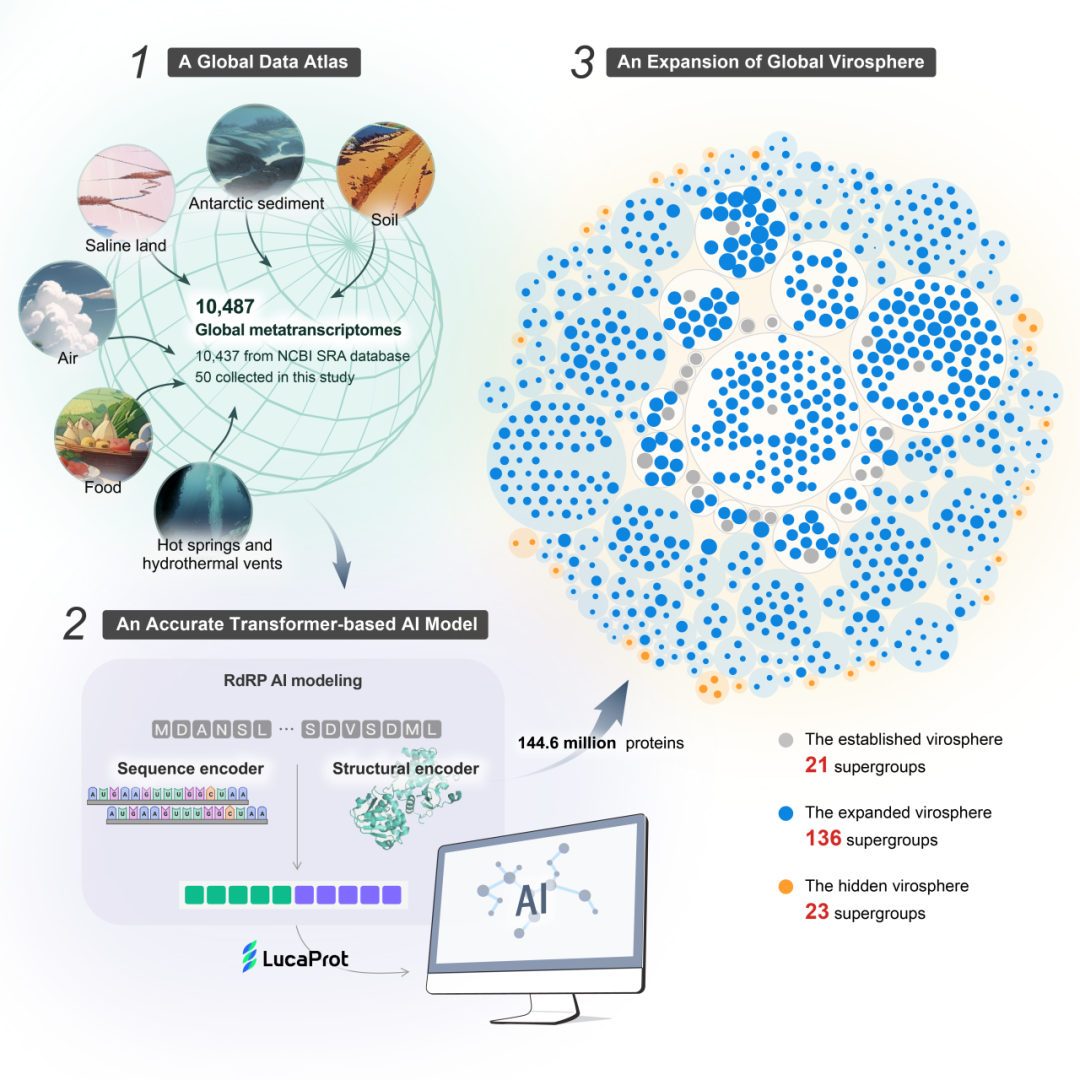
根据介绍,LucaProt 是一种能够进行深度学习的 Transformer 模型,在学习了大量病毒和非病毒基因组序列后,能够自主形成对病毒的判断标准,从而在海量 RNA 测序数据中进行病毒序列的挖掘。在测试中,LucaProt 显示出极高的准确性和特异性,假阳性率为 0.014%,假阴性率为 1.72%。在与其他病毒挖掘工具的比较中,它在处理较长序列时表现出更大的优势。
应用 LucaProt,研究团队对全球生物环境样本中的 10,487 份 RNA 测序数据进行了病毒挖掘,发现了超过 51 万条 病毒基因组,代表了超过 16 万个潜在病毒种类以及 180 个 RNA 病毒超群(相当于门或纲的分类级别), RNA 病毒超群数量扩大约 9 倍。其中 23 个超群无法通过序列同源方法识别,被称为病毒圈的“暗物质”。
该研究还报告了迄今最长的 RNA 病毒基因组,长度达到 47,250 个核苷酸;发现的基因组结构超出了以往的认知,展示了 RNA 病毒基因组进化的灵活性;同时识别到多种病毒功能蛋白,尤其是与细菌相关的功能蛋白,这进一步表明还有更多类型的 RNA 噬菌体待探索。
研究指出,新发现的病毒遍布地球的各种生态环境。总体来看,落叶层、湿地、淡水和废水环境中的病毒多样性最高。不过,在南极底泥、深海热泉、活性污泥和盐碱滩等极端环境中,RNA 病毒的多样性和丰度依然不低,甚至在深海热泉的高温环境中,RNA 病毒仍在积极复制。
尽管 LucaProt 是专为 RNA 病毒发现设计的模型,它也具备识别蛋白质序列和隐含结构信息的功能,因此可以用于蛋白质功能的鉴定。在论文中,研究团队将 LucaProt 模型开源,并通过在线平台与全球科学家共享。